




Page suivante: 4 Analyse structurale Niveau précédent: 3 Cartographie Page précédente: 3.2.1 Cartes de restriction par





Page suivante: 4 Analyse structurale
Niveau précédent: 3 Cartographie
Page précédente: 3.2.1 Cartes de restriction par
La stratégie des fragments chevauchants consiste dans un premier temps à découper l'ADN du génome en longs fragments (quelques centaines de milliers de bases) présentant des régions de chevauchement. Il s'agit ensuite de replacer ces fragments les uns par rapport aux autres. La détection des fragments chevauchants et la détermination de leurs positions relatives peuvent, par exemple, être effectuées à l'aide de sondes. Une sonde est une séquence d'ADN assez courte permettant d'identifier une région particulière du génome. On dit alors qu'elle s' hybride à cette région. Deux fragments s'hybridant à une même sonde sont probablement chevauchants (Figure 6).
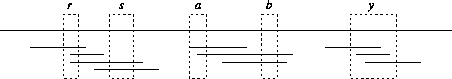
Figure 6: Représentation schématique de plusieurs hybridations. Le génome
est représenté par une ligne noire horizontale. Les fragments
chevauchants sont représentés en dessous par des lignes horizontales
plus courtes. Les sondes sont représentées par les parallépipèdes en
pointillés et indiquent les régions explorées par les sondes. Les
sondes r et s sont reliées par un fragment et doivent donc être
considérées comme étant voisines sur le génome.
Dans une version simplifiée, sans tenir compte des incertitudes, le problème
peut se formuler de la façon suivante : étant donné un ensemble C de
fragments et deux sous-ensembles disjoints de 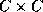 représentant respectivement les paires de fragments qui doivent se
recouvrir et qui ne doivent pas se recouvrir, trouver un ordre des fragments
satisfaisant ces contraintes de recouvrement. Le problème de décision
associé est NP-complet [ Gol 93] et s'interprète
naturellement en terme de graphe d'intervalles. Une modélisation plus
réaliste du problème consisterait, à partir des probabilités de recouvrement
de chaque paire, à déterminer les ordres de vraisemblance maximum.
représentant respectivement les paires de fragments qui doivent se
recouvrir et qui ne doivent pas se recouvrir, trouver un ordre des fragments
satisfaisant ces contraintes de recouvrement. Le problème de décision
associé est NP-complet [ Gol 93] et s'interprète
naturellement en terme de graphe d'intervalles. Une modélisation plus
réaliste du problème consisterait, à partir des probabilités de recouvrement
de chaque paire, à déterminer les ordres de vraisemblance maximum.
Dans [ Cla 94], le problème est abordé via le langage de programmation par contraintes ElipSys, une version parallélisable du langage de programmation par contrainte Eclipse, lui-même issu de Chip, et s'appuyant sur des algorithmes traditionnels du monde CSP. Les données sont issues d'hybridations des fragments avec un ensemble de sondes. Les auteurs procèdent en deux étapes :
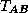 avec lesquels les deux sondes se sont
hybridées ;
avec lesquels les deux sondes se sont
hybridées ;
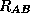 avec lesquels seulement l'une des 2
sondes s'est hybridée (plus
avec lesquels seulement l'une des 2
sondes s'est hybridée (plus 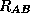 est faible vis-à-vis de
est faible vis-à-vis de 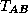 ,
plus il est vraisemblable que les deux sondes soient proches) ;
,
plus il est vraisemblable que les deux sondes soient proches) ;
La modélisation choisie pour représenter ce problème dans le cadre CSP est la suivante :
L'effet positif de l'utilisation des contraintes pour réduire l'espace de recherche dans la phase de résolution est mis en évidence.
Un problème majeur dans l'ordonnancement des fragments chevauchants est le << bruit >> expérimental. Celui-ci a plusieurs sources :





Page suivante: 4 Analyse structurale
Niveau précédent: 3 Cartographie
Page précédente: 3.2.1 Cartes de restriction par
Copyright(C)1995
INRA
Tous droits réservés