




Page suivante: 3.2 Cartographie physique Niveau précédent: 3 Cartographie Page précédente: 3 Cartographie





Page suivante: 3.2 Cartographie physique
Niveau précédent: 3 Cartographie
Page précédente: 3 Cartographie
Les cartes génétiques reposent sur l'évaluation de la distance relative
séparant des caractères héréditaires ou marqueurs génétiques. Un marqueur
génétique est transmis selon les lois de la génétique. La distance génétique
entre deux gènes se définit par la fréquence d'apparition d'événements
de recombinaisons entre ces gènes d'une génération à l'autre. Cette
fréquence de recombinaison constitue l'unité de mesure des cartes
génétiques, 1% de recombinaison correspondant à une distance de 1
centi-Morgan. En reprenant l'exemple de la Figure 1, si 1
% des descendants issus de l'union des deux parents
(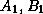 )
)  (
(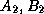 ) sont de type recombinant (
) sont de type recombinant (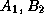 ) ou
(
) ou
(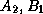 ), alors la distance entre les gènes A et B est de 1
centi-Morgan. La probabilité d'événement de recombinaison entre deux
marqueurs est d'autant plus grande qu'ils sont éloignés sur le chromosome
qui les porte. Une carte génétique représente alors l'ordre relatif des
marqueurs génétiques et les distances les séparant les uns
des autres le long du chromosome.
), alors la distance entre les gènes A et B est de 1
centi-Morgan. La probabilité d'événement de recombinaison entre deux
marqueurs est d'autant plus grande qu'ils sont éloignés sur le chromosome
qui les porte. Une carte génétique représente alors l'ordre relatif des
marqueurs génétiques et les distances les séparant les uns
des autres le long du chromosome.
L'approche dite du maximum de vraisemblance est l'approche statistique
classique utilisée pour estimer les distances entre m marqueurs. La
vraisemblance est la probabilité d'observer une série de valeurs dans un
échantillon étant donnée une carte fixée. Pour un ordre de marqueurs donné
a priori, les distances sont estimées par le vecteur de valeurs (les
distances) qui maximise la vraisemblance de cette carte pour les
recombinaisons observées dans un échantillon de descendants issus de
croisements connus. Les estimations sont calculées par un algorithme
d'optimisation dans 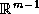 . Le problème reste de trouver l'ordre des
marqueurs dans la carte. Lorsque le nombre de marqueurs est faible (moins de
7) une méthode consiste à calculer la vraisemblance de tous les ordres
possibles (
. Le problème reste de trouver l'ordre des
marqueurs dans la carte. Lorsque le nombre de marqueurs est faible (moins de
7) une méthode consiste à calculer la vraisemblance de tous les ordres
possibles (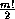 ) et à conserver l'ordre ayant le maximum de
vraisemblance le plus élevé.
) et à conserver l'ordre ayant le maximum de
vraisemblance le plus élevé.
Le système CPROP développé par Letovsky et Berlin [ Let 92] se situe en aval d'éventuels calculs de vraisemblance et est destiné a fusionner des données d'origine diverses : distances issues de l'approche statistique du maximum de vraisemblance, mais aussi des données provenant de cartes physiques par exemple (cartes de restriction, cf. infra). Bien que le système ait été développé pour aider le biologiste dans la construction de la carte génétique de la bactérie Escherichia coli, il est suffisamment générique pour s'appliquer à la cartographie génétique de n'importe quel génome. En effet, partant du constat que toutes les données expérimentales utilisées dans la cartographie génétique peuvent se ramener soit à des distances entre marqueurs, soit à des orientations entre marqueurs, Letovsky et Berlin ont construit CPROP pour traiter tout problème pouvant se ramener à ces deux types de contraintes.
La formulation choisie conduit au CSP suivant :
CPROP utilise des règles d'inférence pour produire de nouvelles contraintes à partir de celles déjà présentes dans le système ou pour réunir des sous-ensembles de marqueurs non disjoints. Si par exemple dans un ensemble 1 on a les marqueurs A, B et C, avec A avant B avant C, et dans un ensemble 2 on a D avant C avant B, on peut déduire que les ensembles 1 et 2 sont orientés en sens inverse et on peut les unir en un seul ensemble où l'on aura A avant B avant C avant D (ou l'inverse). Toutes les inférences produites à partir des autres règles reviennent à faire de la fermeture transitive dans un réseau de contraintes temporelles où la séquence chromosomique serait l'axe du temps. Le résultat est un système clos pour cet ensemble de règles.
Pendant la phase d'inférence, un système de maintien de justifications est mis à jour pour, en cas de contradiction, essayer d'en expliquer les causes au biologiste. Si le réseau est cohérent, une phase de << réduction de l'ensemble des contraintes >> est lancée, pour éliminer les contraintes trivialement redondantes, qui par leur nombre rendraient illisible le résultat fourni. Car contrairement aux systèmes s'appuyant uniquement sur l'approche du maximum de vraisemblance, CPROP ne produit pas un ensemble d'ordres totaux les plus vraisemblables entre marqueurs. Letovsky et Berlyn estiment en effet plus lisible et plus fidèle de donner un ensemble de contraintes qui illustrent les incertitudes restantes. La solution fournie est donc seulement un ordre partiel.





Page suivante: 3.2 Cartographie physique
Niveau précédent: 3 Cartographie
Page précédente: 3 Cartographie
Copyright(C)1995
INRA
Tous droits réservés