




Page suivante: 4.1.2 Visualisation de structures secondaires Niveau précédent: 4.1 Détermination et visualisation de Page précédente: 4.1 Détermination et visualisation de





Page suivante: 4.1.2 Visualisation de structures secondaires
Niveau précédent: 4.1 Détermination et visualisation de
Page précédente: 4.1 Détermination et visualisation de
Le problème de la détermination d'une structure secondaire se traite
différemment selon que l'on dispose d'une seule séquence (une molécule d'ARN
d'un organisme) ou de plusieurs séquences homologues (un ensemble de
molécules d'ARN de même fonction mais appartenant à des organismes
différents). Nous considérons ici le cas où une seule séquence est
disponible. Un inconvénient majeur des approches traditionnelles est la
difficulté à prendre en compte les contraintes de repliement issues de
données expérimentales (imposer un appariement, un non-appariement...)
autoriser localement la présence de pseudo-n ud (cf. infra)...
L'intérêt de l'approche proposée dans [ Gas 94] est double :
permettre à l'utilisateur de spécifier pas à pas une structure hypothétique
par adjonction/retrait de contraintes structurales ; générer l'ensemble des
structures secondaires satisfaisant les contraintes.
ud (cf. infra)...
L'intérêt de l'approche proposée dans [ Gas 94] est double :
permettre à l'utilisateur de spécifier pas à pas une structure hypothétique
par adjonction/retrait de contraintes structurales ; générer l'ensemble des
structures secondaires satisfaisant les contraintes.
Le problème est formulé dans le cadre CSP de la manière suivante :
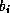 (i désigne le numéro
d'ordre de la base dans la séquence de longueur N) est associée une
variable
(i désigne le numéro
d'ordre de la base dans la séquence de longueur N) est associée une
variable 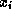 ;
;
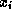 prend ses valeurs dans
l'intervalle d'entiers
prend ses valeurs dans
l'intervalle d'entiers 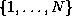 , chaque valeur j de ce domaine
représentant un partenaire possible
, chaque valeur j de ce domaine
représentant un partenaire possible 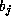 de
de 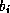 dans une structure
secondaire ; les bases n'étant pas toutes appariées dans une structure
secondaire, la valeur i dans le domaine de
dans une structure
secondaire ; les bases n'étant pas toutes appariées dans une structure
secondaire, la valeur i dans le domaine de 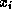 représente le fait que
la base
représente le fait que
la base 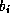 n'est pas appariée dans la structure solution ;
n'est pas appariée dans la structure solution ;
 uds (formellement, deux paires de bases
uds (formellement, deux paires de bases
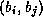 et
et 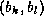 telles que i<j et k<l forment un
pseudo-n
telles que i<j et k<l forment un
pseudo-n ud si et seulement si i<k<j<l ou k<i<l<j) ;
ud si et seulement si i<k<j<l ou k<i<l<j) ;
Une fois le problème posé, le nombre de solutions satisfaisant
les contraintes
reste de l'ordre de 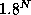 (N représente la taille de la séquence).
Les auteurs ont ajouté quelques contraintes supplémentaires
qui permettent de
réduire considérablement l'espace de recherche en faisant varier la taille
minimum des hélices et en prenant en compte la stabilité de la molécule
(sous forme de probabilités d'appariement) et les données expérimentales.
Dans une première étape, dite de spécification, le maintien d'arc
cohérence est recherché.
(N représente la taille de la séquence).
Les auteurs ont ajouté quelques contraintes supplémentaires
qui permettent de
réduire considérablement l'espace de recherche en faisant varier la taille
minimum des hélices et en prenant en compte la stabilité de la molécule
(sous forme de probabilités d'appariement) et les données expérimentales.
Dans une première étape, dite de spécification, le maintien d'arc
cohérence est recherché.
Une fois les propriétés de la structure secondaire spécifiées,
un algorithme de recherche de
cliques maximales travaille sur un sous-graphe de la
micro-structure du
réseau de contraintes pour produire l'ensemble des structures secondaires
saturées (maximales au sens de l'inclusion sur les ensembles d'appariements)
satisfaisant l'ensemble des contraintes.
du
réseau de contraintes pour produire l'ensemble des structures secondaires
saturées (maximales au sens de l'inclusion sur les ensembles d'appariements)
satisfaisant l'ensemble des contraintes.





Page suivante: 4.1.2 Visualisation de structures secondaires
Niveau précédent: 4.1 Détermination et visualisation de
Page précédente: 4.1 Détermination et visualisation de
Copyright(C)1995
INRA
Tous droits réservés