




Page suivante: Détermination d'un modèle consensus
Niveau précédent: 4.1.3 Détermination de structures tertiaires
Page précédente: 4.1.3 Détermination de structures tertiaires
Dans MC-SYM [ Maj 91], les auteurs s'appuient sur la structure
secondaire et les données expérimentales pour spécifier les propriétés du
modèle tridimensionnel recherché. La formulation dans le cadre CSP peut se
décrire comme suit :
- variables :
- à chaque nucléotide est associée une variable ;
- valeurs :
- ce sont les conformations spatiales autorisées du
nucléotide ; sachant qu'une telle conformation est liée à sept angles de
rotation (définissant ainsi un très grand nombre de conformations
possibles), les auteurs proposent de calculer un domaine restreint de
valeurs en s'appuyant sur un ensemble statistiquement établi de
conformations spatiales (atomiques) connues ;
- contraintes :
- un indicateur conformationnel, associé à chaque
nucléotide, décrit les relations spatiales entre ce nucléotide et les
autres ; c'est un moyen d'incorporer des données spécifiques à la molécule
étudiée ; à chaque indicateur est associée une fonction permettant de
générer les valeurs possibles de la variable associée au nucléotide (les
contraintes unaires sont encodées dans ce descripteur, en quelque sorte) ;
les autres contraintes se ramènent à des contraintes de distances
(égalités/inégalités) ; par exemple, contrainte de distance pour une
boucle << d'épingle à cheveux >> à 3 nucléotides (
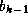 ,
, 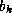 ,
,
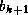 ) qui doit fermer sur sa base d'hélice.
) qui doit fermer sur sa base d'hélice.
Cette approche est encore limitée à des séquences de petite taille et
l'importance de la connaissance des interactions 3D y est cruciale pour
obtenir des modèles << faisables >> et corrects.
Auteurs:Christine Gaspin, Christian Bessiere, Annick Moisan et Thomas Schiex
Dernière mise à jour: jeudi, 11 janvier 1996, 18:28:04 MET
Institut National de la Recherche Agronomique
Département de Biométrie et Intelligence Artificielle
Copyright(C)1995
INRA
Tous droits réservés










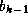 ,
, 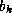 ,
,
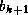 ) qui doit fermer sur sa base d'hélice.
) qui doit fermer sur sa base d'hélice.